2. Traitement de données avec PANDAS#
Marc BUFFAT, Université Claude Bernard Lyon 1

%matplotlib inline
from IPython.display import HTML,display
# bibliotheques de base
import matplotlib
import numpy as np
import matplotlib.pyplot as plt
from matplotlib import rcParams
rcParams['font.family'] = 'serif'
rcParams['font.size'] = 14
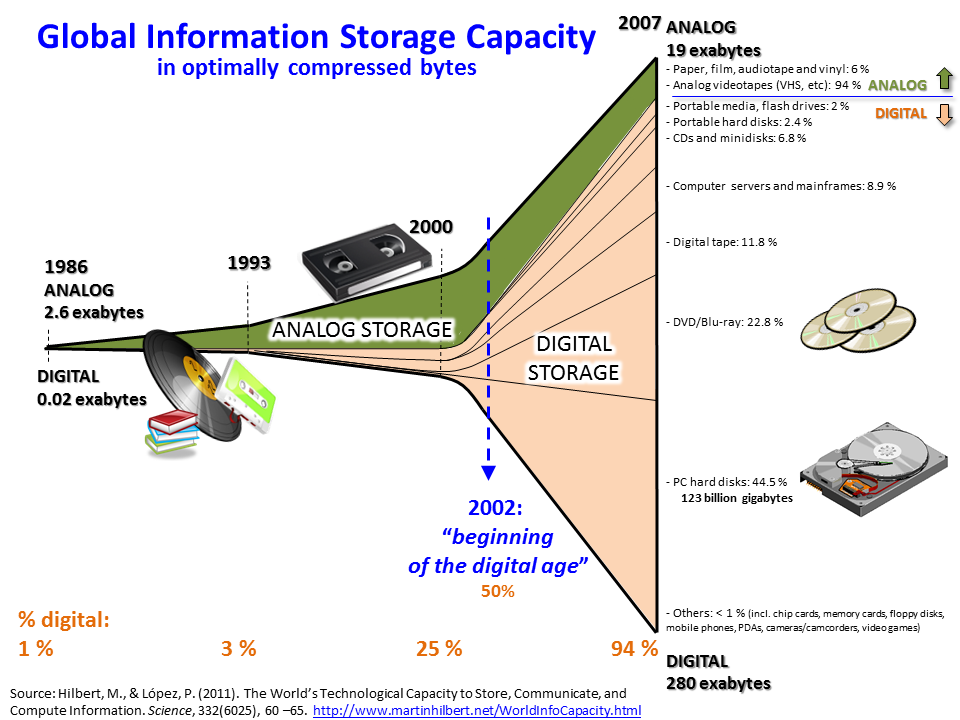
2.1. BIG DATA#

2.2. croissance du stocage digital#
2.3. Les bons outils suivant la taille des données#
petits fichiers ( ~ qques ko) : tableur: excel locacl
1 Mo < x < 100 Mo : très lourd avec excel => script: Python (Pandas), R
100 Mo < xx < 100 Go : impossible avec excel ==> script: Python (Pandas), R, SQL
100 Go < xxx < 1 To ===> SQL base de données
1 Tb et + ====> Hadoop
2.4. Traitement des données#
2.4.1. the (old) excel way#
utilisation d’un tableur (excel , localc)
simple pour des données simples
mais tricky pour des traitements complexes (VBA)
2.4.2. the (new) Pandas way#
basé sur un vrai language de programmation (Python)
traitement complexe des données (cleaning,..)
automatisation
performance
adapté au big data
2.5. Pandas: « Python Library for Data Analysis and Statistics »#

2.5.1. Pandas: http://pandas.pydata.org#
Pandas (from Panel Data analysis) is a Python package providing fast, flexible, and expressive data structures designed to work with relational or labeled data both. It is a fundamental high-level building block for doing practical, real world data analysis in Python.
pandas is well suited for:
Tabular data with heterogeneously-typed columns, as in an SQL table or Excel spreadsheet
Ordered and unordered (not necessarily fixed-frequency) time series data.
Arbitrary matrix data (homogeneously typed or heterogeneous) with row and column labels
Any other form of observational / statistical data sets. The data actually need not be labeled at all to be placed into a pandas data structure
Exemples
2.5.2. Key features:#
Easy handling of missing data
Size mutability: columns can be inserted and deleted from DataFrame and higher dimensional objects
Automatic and explicit data alignment: objects can be explicitly aligned to a set of labels, or the data can be aligned automatically
Powerful, flexible group by functionality to perform split-apply-combine operations on data sets
Intelligent label-based slicing, fancy indexing, and subsetting of large data sets
Intuitive merging and joining data sets
Flexible reshaping and pivoting of data sets
Hierarchical labeling of axes
Robust IO tools for loading data from flat files, Excel files, databases, and HDF5
Time series functionality: date range generation and frequency conversion, moving window statistics, moving window linear regressions, date shifting and lagging, etc.
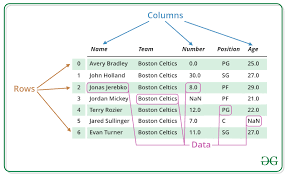
2.6. Data structure in Pandas#
Type de données de base: dataframe (tableau de type excel)
Une DataFrame PANDAS est une structure de données tabulaire bidimensionnelle de taille variable, potentiellement hétérogène, avec des axes libellés (lignes et colonnes). est une structure de données bidimensionnelle, c’est-à-dire que les données sont alignées de manière tabulaire en rangées et en colonnes.
import pandas as pd
2.7. DataFrame#
structure 2D équivalente à un tableur
structure hétérogéne avec index (ligne) et column (colonnes)
2.7.1. création à partir de tableaux numpy#
n=5
d = np.ones((n,2))
pd.DataFrame(d)
| 0 | 1 | |
|---|---|---|
| 0 | 1.0 | 1.0 |
| 1 | 1.0 | 1.0 |
| 2 | 1.0 | 1.0 |
| 3 | 1.0 | 1.0 |
| 4 | 1.0 | 1.0 |
2.7.2. création à partir de dictionnaire et de tableaux#
2.7.2.1. nom de colonnes#
n = 5
d = { 'One' : np.linspace(1,n,n), 'Two': np.linspace(1,n,n)**2}
pd.DataFrame(d)
| One | Two | |
|---|---|---|
| 0 | 1.0 | 1.0 |
| 1 | 2.0 | 4.0 |
| 2 | 3.0 | 9.0 |
| 3 | 4.0 | 16.0 |
| 4 | 5.0 | 25.0 |
2.7.2.2. nom de ligne (index)#
data=pd.DataFrame(d,index=['a','b','c','d','e'])
data
| One | Two | |
|---|---|---|
| a | 1.0 | 1.0 |
| b | 2.0 | 4.0 |
| c | 3.0 | 9.0 |
| d | 4.0 | 16.0 |
| e | 5.0 | 25.0 |
2.7.2.3. selection colonne#
data['Two']
a 1.0
b 4.0
c 9.0
d 16.0
e 25.0
Name: Two, dtype: float64
data['Two']['b']
4.0
data['Two'][1]
4.0
2.7.2.4. selection ligne#
data.loc['a']['One']
1.0
data.loc['a']
One 1.0
Two 1.0
Name: a, dtype: float64
data.loc['a']['Two']
1.0
data.loc['a'][0]
1.0
# position ligne/colonne
data.iloc[2,1]
9.0
data.iloc[0,0]
1.0
2.8. DataFrame à partir de fichier csv#
fichier csv: Comma-separated values, connu sous le sigle CSV, est un format informatique ouvert représentant des données tabulaires sous forme de valeurs séparées par des virgules dans un fichier texte. C’est un format d’échange courant pour les tableurs.
variante française: chiffre avec virgule, séparateur ;
2.8.1. lecture de données issue de Hubble#
le nom des colonnes est dans le fichier csv et l’index des lignes est numérique (de 0 a n-1)
!head hubble_data.csv
distance,recession_velocity
.032,170
.034,290
.214,-130
.263,-70
.275,-185
.275,-220
.45,200
.5,290
.5,270
data = pd.read_csv("hubble_data.csv")
data.head()
| distance | recession_velocity | |
|---|---|---|
| 0 | 0.032 | 170 |
| 1 | 0.034 | 290 |
| 2 | 0.214 | -130 |
| 3 | 0.263 | -70 |
| 4 | 0.275 | -185 |
data.info()
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 24 entries, 0 to 23
Data columns (total 2 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 distance 24 non-null float64
1 recession_velocity 24 non-null int64
dtypes: float64(1), int64(1)
memory usage: 512.0 bytes
2.8.1.1. change le nom des colonnes#
data.columns = ['dist','rec_vel']
data.tail()
| dist | rec_vel | |
|---|---|---|
| 19 | 1.7 | 960 |
| 20 | 2.0 | 500 |
| 21 | 2.0 | 850 |
| 22 | 2.0 | 800 |
| 23 | 2.0 | 1090 |
2.8.1.2. lecture fichier csv sans entete de noms de colonnes#
data = pd.read_csv("hubble_data_no_headers.csv",
names=['dist','rec_vel'])
data.tail()
| dist | rec_vel | |
|---|---|---|
| 19 | 1.7 | 960 |
| 20 | 2.0 | 500 |
| 21 | 2.0 | 850 |
| 22 | 2.0 | 800 |
| 23 | 2.0 | 1090 |
2.8.2. accés aux données#
data.info()
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 24 entries, 0 to 23
Data columns (total 2 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 dist 24 non-null float64
1 rec_vel 24 non-null int64
dtypes: float64(1), int64(1)
memory usage: 512.0 bytes
2.8.2.1. selection colonne#
data['dist'].tail()
19 1.7
20 2.0
21 2.0
22 2.0
23 2.0
Name: dist, dtype: float64
2.8.2.2. sélection de ligne dans la colonne#
data['dist'][0:5]
0 0.032
1 0.034
2 0.214
3 0.263
4 0.275
Name: dist, dtype: float64
2.8.2.3. sélection de plusieurs colonnes#
data[['rec_vel','dist','rec_vel']].head()
| rec_vel | dist | rec_vel | |
|---|---|---|---|
| 0 | 170 | 0.032 | 170 |
| 1 | 290 | 0.034 | 290 |
| 2 | -130 | 0.214 | -130 |
| 3 | -70 | 0.263 | -70 |
| 4 | -185 | 0.275 | -185 |
2.8.3. calcul sur les données#
2.8.3.1. calcul de l’énergie: \(E = K*dist + 0.5 *v^2\)#
K = 1000.
data['E'] = K*data['dist'] + 0.5*data['rec_vel']**2
data['dist2'] = data['dist']**2
data.head()
| dist | rec_vel | E | dist2 | |
|---|---|---|---|---|
| 0 | 0.032 | 170 | 14482.0 | 0.001024 |
| 1 | 0.034 | 290 | 42084.0 | 0.001156 |
| 2 | 0.214 | -130 | 8664.0 | 0.045796 |
| 3 | 0.263 | -70 | 2713.0 | 0.069169 |
| 4 | 0.275 | -185 | 17387.5 | 0.075625 |
2.8.3.2. suppression d’une colonne#
del data['dist2']
data.head()
| dist | rec_vel | E | |
|---|---|---|---|
| 0 | 0.032 | 170 | 14482.0 |
| 1 | 0.034 | 290 | 42084.0 |
| 2 | 0.214 | -130 | 8664.0 |
| 3 | 0.263 | -70 | 2713.0 |
| 4 | 0.275 | -185 | 17387.5 |
2.8.3.3. suppression d’un ligne (attention paramêtre inplace)#
data.drop(1)
| dist | rec_vel | E | |
|---|---|---|---|
| 0 | 0.032 | 170 | 14482.0 |
| 2 | 0.214 | -130 | 8664.0 |
| 3 | 0.263 | -70 | 2713.0 |
| 4 | 0.275 | -185 | 17387.5 |
| 5 | 0.275 | -220 | 24475.0 |
| 6 | 0.450 | 200 | 20450.0 |
| 7 | 0.500 | 290 | 42550.0 |
| 8 | 0.500 | 270 | 36950.0 |
| 9 | 0.630 | 200 | 20630.0 |
| 10 | 0.800 | 300 | 45800.0 |
| 11 | 0.900 | -30 | 1350.0 |
| 12 | 0.900 | 650 | 212150.0 |
| 13 | 0.900 | 150 | 12150.0 |
| 14 | 0.900 | 500 | 125900.0 |
| 15 | 1.000 | 920 | 424200.0 |
| 16 | 1.100 | 450 | 102350.0 |
| 17 | 1.100 | 500 | 126100.0 |
| 18 | 1.400 | 500 | 126400.0 |
| 19 | 1.700 | 960 | 462500.0 |
| 20 | 2.000 | 500 | 127000.0 |
| 21 | 2.000 | 850 | 363250.0 |
| 22 | 2.000 | 800 | 322000.0 |
| 23 | 2.000 | 1090 | 596050.0 |
data.head()
| dist | rec_vel | E | |
|---|---|---|---|
| 0 | 0.032 | 170 | 14482.0 |
| 1 | 0.034 | 290 | 42084.0 |
| 2 | 0.214 | -130 | 8664.0 |
| 3 | 0.263 | -70 | 2713.0 |
| 4 | 0.275 | -185 | 17387.5 |
data.iloc[1,0]
0.034
data.drop(1,inplace=True)
data.head()
| dist | rec_vel | E | |
|---|---|---|---|
| 0 | 0.032 | 170 | 14482.0 |
| 2 | 0.214 | -130 | 8664.0 |
| 3 | 0.263 | -70 | 2713.0 |
| 4 | 0.275 | -185 | 17387.5 |
| 5 | 0.275 | -220 | 24475.0 |
2.8.4. indexation des données:#
Au lieu d’utiliser l’indexation numérique de 0 à n-1, on va indexer les données par rapport à une colonne, par ex. la colonne “dist” pour annalyser
vel = F(dist)
E = G(dist)
data.set_index("dist",inplace=True)
data.head()
| rec_vel | E | |
|---|---|---|
| dist | ||
| 0.032 | 170 | 14482.0 |
| 0.214 | -130 | 8664.0 |
| 0.263 | -70 | 2713.0 |
| 0.275 | -185 | 17387.5 |
| 0.275 | -220 | 24475.0 |
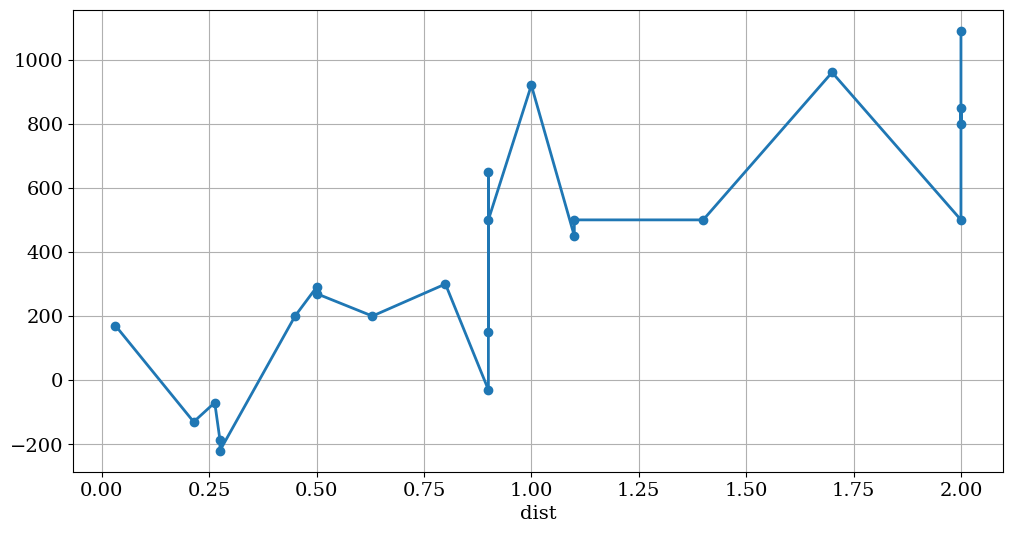
2.8.4.1. tracer des données rec_vel = F(dist)#
data['rec_vel'].plot(figsize=(12,6),lw=2,grid=True,style='-o')
plt.title("")
Text(0.5, 1.0, '')
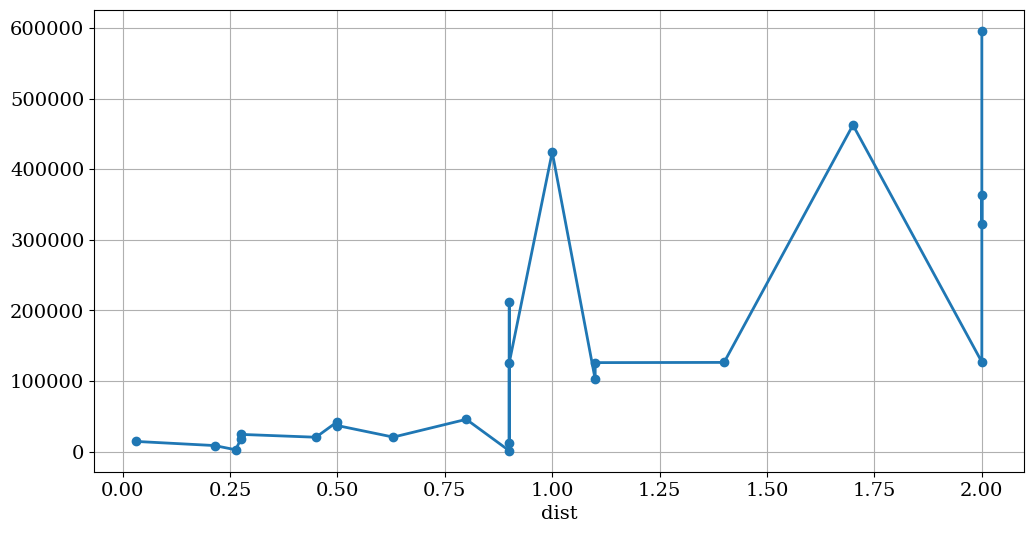
data['E'].plot(figsize=(12,6),lw=2,grid=True,style='-o')
<Axes: xlabel='dist'>
2.8.4.2. traitement statistique:#
moyenne , medianne …
analyse par colonne
energy = data['E']
type(energy)
pandas.core.series.Series
energy.mean(), energy.median()
(140673.97826086957, 45800.0)
analyse globale
data.describe()
| rec_vel | E | |
|---|---|---|
| count | 23.000000 | 23.000000 |
| mean | 376.739130 | 140673.978261 |
| std | 379.166576 | 172733.702836 |
| min | -220.000000 | 1350.000000 |
| 25% | 160.000000 | 18918.750000 |
| 50% | 300.000000 | 45800.000000 |
| 75% | 575.000000 | 169575.000000 |
| max | 1090.000000 | 596050.000000 |
2.8.5. analyse statistique#
variance, covariance, moyenne …..
matrice de covariance entre 3 tableaux x,y,z
avec (\(E\) est l’opérateur moyenne \(E(X) = \bar{X}\)) $\( cov_{x,y} = E[(X – E[X])(Y – E[Y])] = \overline{(X-\bar{X})(Y-\bar{Y})} = \frac{\sum(X_i – \bar{X})(Y_i – \bar{Y})}{N} \)$
data1 = data.reset_index()
data1.head()
| dist | rec_vel | E | |
|---|---|---|---|
| 0 | 0.032 | 170 | 14482.0 |
| 1 | 0.214 | -130 | 8664.0 |
| 2 | 0.263 | -70 | 2713.0 |
| 3 | 0.275 | -185 | 17387.5 |
| 4 | 0.275 | -220 | 24475.0 |
CV = data1.cov()
display(CV)
| dist | rec_vel | E | |
|---|---|---|---|
| dist | 0.399092 | 1.943741e+02 | 8.239335e+04 |
| rec_vel | 194.374051 | 1.437673e+05 | 5.939248e+07 |
| E | 82393.346148 | 5.939248e+07 | 2.983693e+10 |
# CV est un datafrme : accès
CV['rec_vel']['dist'] , CV.iloc[0,1]
(194.37405138339918, 194.37405138339918)
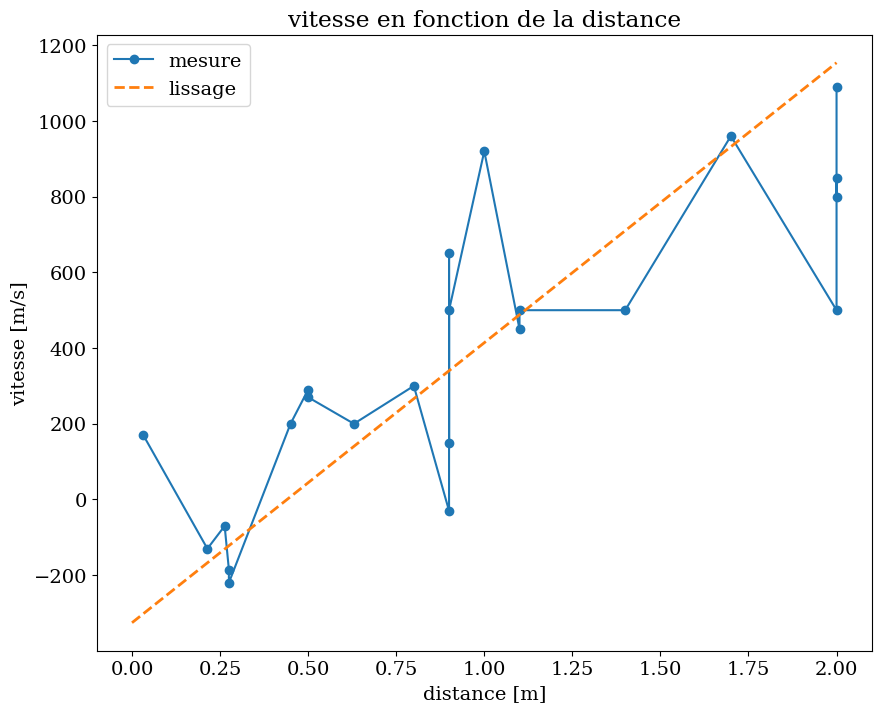
vérification: lissage par droite moindre carré V=F(dist)
avec \( a = \frac{\sigma_{V,V}}{\sigma_{V,dist}} \) et \( b = \bar{V} - a * \overline{dist} \)
# verification lissage par droite moindre carré V=F(dist)
a = CV.iloc[1,1]/CV.iloc[1,0]
b = data1['rec_vel'].mean() - a * data1['dist'].mean()
print(a,b)
739.6424135160937 -325.5674203816508
rcParams['font.family'] = 'serif'
rcParams['font.size'] = 14
X = np.linspace(0.,2.,21)
plt.figure(figsize=(10,8))
plt.plot(data1['dist'],data1['rec_vel'],'-o',label="mesure")
plt.plot(X,a*X+b,'--',lw=2,label="lissage")
plt.legend()
plt.xlabel('distance [m]')
plt.ylabel('vitesse [m/s]')
plt.title("vitesse en fonction de la distance")
Text(0.5, 1.0, 'vitesse en fonction de la distance')
2.8.6. modification des données (loc, iloc)#
attention à l’aliasing !!! de nombreuses fonctions renvoient une copy du tableau (paramêtre inplace)
data['E']
dist
0.032 14482.0
0.214 8664.0
0.263 2713.0
0.275 17387.5
0.275 24475.0
0.450 20450.0
0.500 42550.0
0.500 36950.0
0.630 20630.0
0.800 45800.0
0.900 1350.0
0.900 212150.0
0.900 12150.0
0.900 125900.0
1.000 424200.0
1.100 102350.0
1.100 126100.0
1.400 126400.0
1.700 462500.0
2.000 127000.0
2.000 363250.0
2.000 322000.0
2.000 596050.0
Name: E, dtype: float64
data['E'][2.]=1620
print(data.tail())
rec_vel E
dist
1.7 960 462500.0
2.0 500 1620.0
2.0 850 1620.0
2.0 800 1620.0
2.0 1090 1620.0
/tmp/ipykernel_649710/1408120798.py:1: SettingWithCopyWarning:
A value is trying to be set on a copy of a slice from a DataFrame
See the caveats in the documentation: https://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy
data['E'][2.]=1620
2.8.6.1. utilisation de .at (attention ligne, colonne)#
data.at[2.,'E']=1800
print(data.tail())
rec_vel E
dist
1.7 960 462500.0
2.0 500 1800.0
2.0 850 1800.0
2.0 800 1800.0
2.0 1090 1800.0
data.loc[2,'E']
dist
2.0 1800.0
2.0 1800.0
2.0 1800.0
2.0 1800.0
Name: E, dtype: float64
2.8.7. autres structures de données#
Serie 1D
xarray: multi-dimensionnal
2.9. DataFrame a partir de fichier csv (exemple 2)#
data = pd.read_csv("wages_hours.csv")
data.head()
| HRS\tRATE\tERSP\tERNO\tNEIN\tASSET\tAGE\tDEP\tRACE\tSCHOOL | |
|---|---|
| 0 | 2157\t2.905\t1121\t291\t380\t7250\t38.5\t2.340... |
| 1 | 2174\t2.970\t1128\t301\t398\t7744\t39.3\t2.335... |
| 2 | 2062\t2.350\t1214\t326\t185\t3068\t40.1\t2.851... |
| 3 | 2111\t2.511\t1203\t49\t117\t1632\t22.4\t1.159\... |
| 4 | 2134\t2.791\t1013\t594\t730\t12710\t57.7\t1.22... |
2.9.1. selection du bon séparateur#
data = pd.read_csv("wages_hours.csv", sep = "\t")
data.head()
| HRS | RATE | ERSP | ERNO | NEIN | ASSET | AGE | DEP | RACE | SCHOOL | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 2157 | 2.905 | 1121 | 291 | 380 | 7250 | 38.5 | 2.340 | 32.1 | 10.5 |
| 1 | 2174 | 2.970 | 1128 | 301 | 398 | 7744 | 39.3 | 2.335 | 31.2 | 10.5 |
| 2 | 2062 | 2.350 | 1214 | 326 | 185 | 3068 | 40.1 | 2.851 | * | 8.9 |
| 3 | 2111 | 2.511 | 1203 | 49 | 117 | 1632 | 22.4 | 1.159 | 27.5 | 11.5 |
| 4 | 2134 | 2.791 | 1013 | 594 | 730 | 12710 | 57.7 | 1.229 | 32.5 | 8.8 |
#pd.read_csv?
2.9.2. Extraction de colonnes a traiter#
data2 = data[["AGE", "RATE"]]
data2.head()
| AGE | RATE | |
|---|---|---|
| 0 | 38.5 | 2.905 |
| 1 | 39.3 | 2.970 |
| 2 | 40.1 | 2.350 |
| 3 | 22.4 | 2.511 |
| 4 | 57.7 | 2.791 |
2.9.3. tri des données#
data_sorted = data2.sort_values("AGE")
data_sorted.head()
| AGE | RATE | |
|---|---|---|
| 3 | 22.4 | 2.511 |
| 27 | 37.2 | 3.015 |
| 31 | 37.4 | 1.901 |
| 33 | 37.5 | 1.899 |
| 32 | 37.5 | 3.009 |
2.9.3.1. indexation en fonction de la colonne AGE#
data_sorted.set_index("AGE", inplace=True)
data_sorted.head()
| RATE | |
|---|---|
| AGE | |
| 22.4 | 2.511 |
| 37.2 | 3.015 |
| 37.4 | 1.901 |
| 37.5 | 1.899 |
| 37.5 | 3.009 |
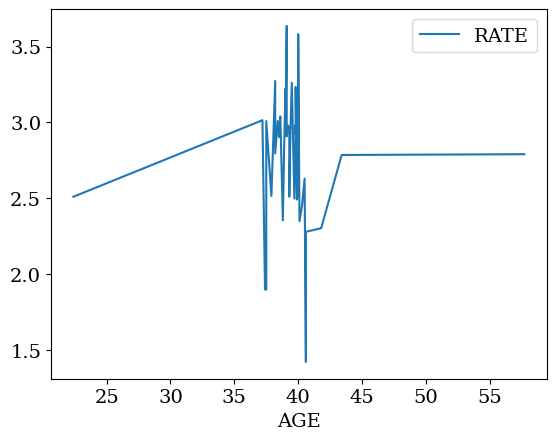
2.9.3.2. tracer du salaire en fonction de l’age#
data_sorted.plot()
plt.show()
2.10. données fonction du temps#
2.10.1. gestion date (conversion)#
date=pd.to_datetime('2016-09-29T23:00:00+02:00')
date
Timestamp('2016-09-29 23:00:00+0200', tz='pytz.FixedOffset(120)')
2.10.2. localisation: timezone#
heure universelle
timezone:
Europe/Paris
date.tz_convert(tz='Europe/Paris')
Timestamp('2016-09-29 23:00:00+0200', tz='Europe/Paris')
2.10.3. serie temporelle (regroupement)#
création de la série temporelle puis du dataframe
date=pd.date_range('2015-01', '2015-12-31',
freq='D')
date
DatetimeIndex(['2015-01-01', '2015-01-02', '2015-01-03', '2015-01-04',
'2015-01-05', '2015-01-06', '2015-01-07', '2015-01-08',
'2015-01-09', '2015-01-10',
...
'2015-12-22', '2015-12-23', '2015-12-24', '2015-12-25',
'2015-12-26', '2015-12-27', '2015-12-28', '2015-12-29',
'2015-12-30', '2015-12-31'],
dtype='datetime64[ns]', length=365, freq='D')
ts = pd.Series(np.random.randn(len(date)),
index=date)
data= ts.to_frame(name='val')
data.head()
| val | |
|---|---|
| 2015-01-01 | -0.752352 |
| 2015-01-02 | -0.986602 |
| 2015-01-03 | -0.411193 |
| 2015-01-04 | 0.723017 |
| 2015-01-05 | -0.702471 |
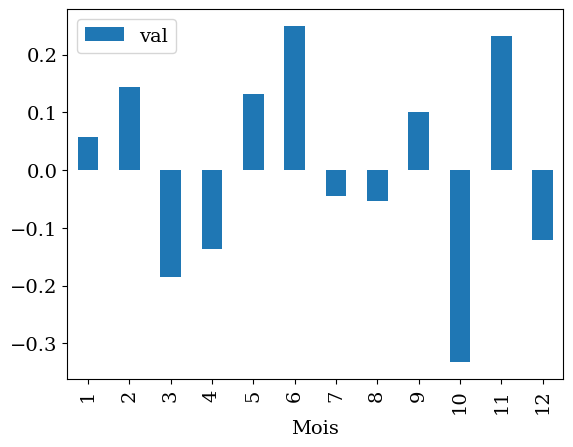
2.10.4. aggregation / mois#
data['Mois']=data.index.month
data.head()
| val | Mois | |
|---|---|---|
| 2015-01-01 | -0.752352 | 1 |
| 2015-01-02 | -0.986602 | 1 |
| 2015-01-03 | -0.411193 | 1 |
| 2015-01-04 | 0.723017 | 1 |
| 2015-01-05 | -0.702471 | 1 |
datam=data.groupby('Mois').aggregate(np.mean)
datam.head()
| val | |
|---|---|
| Mois | |
| 1 | 0.057660 |
| 2 | 0.143202 |
| 3 | -0.185088 |
| 4 | -0.136736 |
| 5 | 0.132138 |
datam.plot(kind='bar')
<Axes: xlabel='Mois'>
2.10.5. boucle sur les valeurs d’une colonne#
somme=0.0
for val in data['val']:
somme += val
print("somme de la colonne :",somme)
somme de la colonne : 0.4795923009327734
2.10.6. avec un index#
data['val2']=0.0
for index,row in data.iterrows():
val = row['val']
data.loc[index,'val2'] = val**2
data.head()
| val | Mois | val2 | |
|---|---|---|---|
| 2015-01-01 | -0.752352 | 1 | 0.566033 |
| 2015-01-02 | -0.986602 | 1 | 0.973384 |
| 2015-01-03 | -0.411193 | 1 | 0.169080 |
| 2015-01-04 | 0.723017 | 1 | 0.522754 |
| 2015-01-05 | -0.702471 | 1 | 0.493465 |
2.11. Bibliothèque spécialisée#
Visualisation de données (statistique)
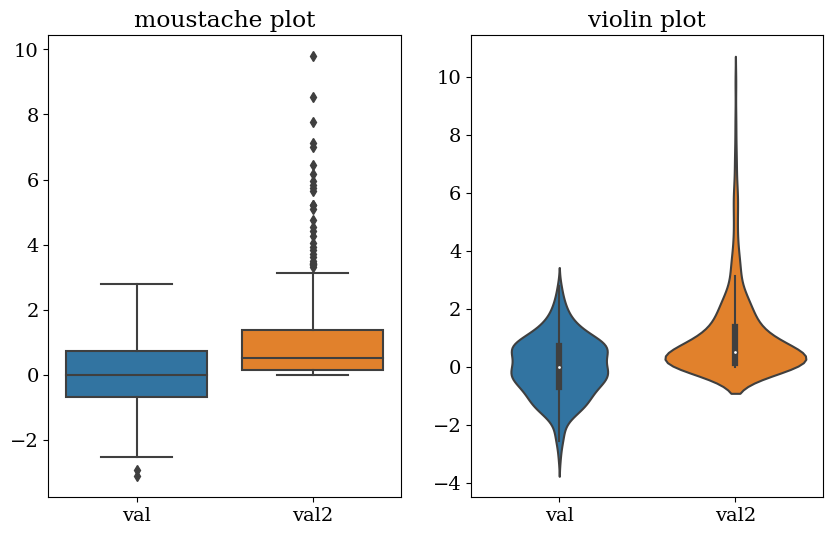
Boîte à moustaches: représentation visuelle permettant mesurer la distribution des données.
Le minimum est affiché à l’extrême gauche du graphique, à la fin de la « moustache » de gauche
Le premier quartile, Q1, est l’extrême gauche de la boîte (moustache gauche)
La médiane est représentée par une ligne au centre de la boîte
Troisième quartile, Q3, affiché à l’extrême droite de la boîte (moustache droite)
Le maximum est à l’extrême droite de la case
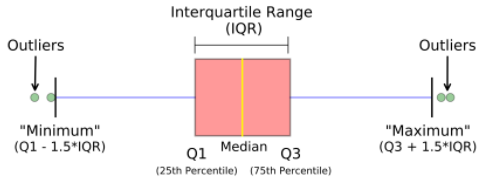
On appelle Écart interquartile ou InterQuartile Range: IQR = Q3-Q1 qui mesure la dispersion des données (estimateur statistique robuste)
Minimum = Q1 - 1.5 IQR
Maximum = Q3 + 1.5 IQR
les points en dehors d’une répartition standard (outliers), i.e en dehors du [minimum:maximum], sont marquées comme des points
violin plot (variante)
Un violin plot est utilisé pour visualiser la distribution des données et sa densité de probabilité.
seaborn: statistical data visualization
import seaborn as sns
# donnees brutes
plt.figure(figsize=(12,6))
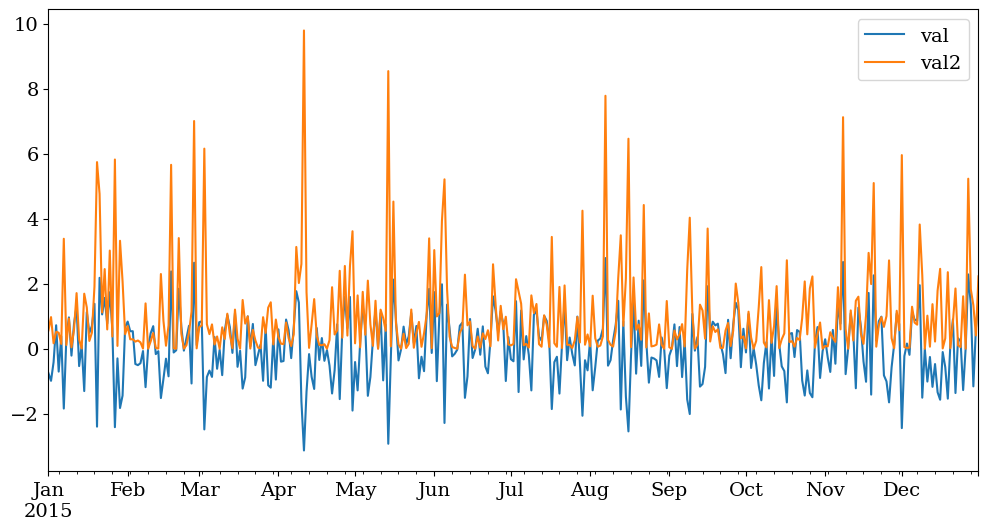
data['val'].plot(label='val')
data['val2'].plot(label='val2')
plt.legend()
<matplotlib.legend.Legend at 0x7f54a8bc9a80>
plt.figure(figsize=(10,6))
plt.subplot(1,2,1)
sns.boxplot(data=data[['val','val2']])
plt.title("moustache plot");
plt.subplot(1,2,2)
sns.violinplot(data=data[['val','val2']])
plt.title("violin plot");
2.12. Bibliographie#
apprendre à lire et à chercher dans la documentation (très (trop) riche !!)
