- dim. 04 février 2018
- Cours
- #ipython jupyter
Table des matières
- 1 Python scientifique (numpy)
- 2 Tableaux avec numpy
- 3 Lecture / Ecriture des données
- 4 Bibliothèque SCIPY
- 5 Tracé avec Matplotlib
- 6 Documentations supplémentaires
- 7 Fin de la leçon
%matplotlib inline
%autosave 300
import numpy as np
import scipy as sp
import matplotlib.pyplot as plt
from matplotlib import rcParams
rcParams['font.family'] = 'serif'
rcParams['font.size'] = 14
from IPython.core.display import HTML
from IPython.display import display
from matplotlib import animation
css_file = 'style.css'
HTML(open(css_file, "r").read())
Python scientifique (numpy)¶
Marc BUFFAT, dpt mécanique, Université Claude Bernard Lyon 1

Mise à disposition selon les termes de la Licence Creative Commons
Attribution - Pas d’Utilisation Commerciale - Partage dans les Mêmes Conditions 2.0 France.
Tableaux avec numpy¶
La bibliothéque Numpy implémente une gestion efficace des tableaux pour représenter les vecteurs et matrices
import numpy as np
- tableau (array): ensemble ordonnée de valeurs de même type (entier, réel) accessible à partir d’un indice commencant à 0. La dimension d’un tableau est fixe.
- liste (list): ensemble ordonnée de valeurs pas forcement du même type. La dimension d’une liste est variable.
Différences entre tableaux et listes¶
# tableau
X=np.array([1.,2,3.0])
X[0]=0
print(" X=",X)
# liste
Y=[1.,2,3.0]
Y[0]=0
print(" Y=",Y)
# ajout d'un élément
Y.append('quatre')
print("Y=",Y)
# dimension de X
print("nbre de dimension =",X.ndim)
print("taille en octets de X = ",X.nbytes," taille elt=",X.itemsize)
# tableau en 2D
X=np.array([[1.,2.],[3.,4]],dtype=np.float64)
print("X=",X)
print("dim X=",X.shape,X.size)
# liste de liste
Y=[[1.,2.],[3,4]]
print("Y=",Y)
print("dim Y=",len(Y),len(Y[0]),len(Y[1]))
Comparaison création¶
On peut représenter des vecteurs et tableaux avec des listes mais c’est très inefficace!!!
# creation tableau de N elts
def creationTab(N):
T=np.zeros((N),dtype=np.int64)
return T
N=10000
%timeit creationTab(N)
# creation liste de N elts
def creationList(N):
L=[0]*N
return L
N=10000
%timeit creationList(N)
Créations de tableaux¶
avec la fonction array
X=np.array(object,dtype=type) X=np.array(list,dtype=type)object = un tableau ou equivalent
list = une liste de valeurs
type = type des elements
int float complex bool np.int32, np.int64, np.float32, np.float54, np.complex128avec des fonction de numpy
- sans initialisation des valeurs
shape = liste des dimensions entre () (tuple)X=np.empty(shape) initialisation valeurs constante nulle ou #0
X=np.zeros(shape,dtype) X=np.ones(shape,dtype)intervalle de valeurs
X=np.arange(deb,fin,pas) X=np.linspace(xdeb,xfin,nb_de_points)meshgrid en dimension >= 2D
X,Y=np.meshgrid(X0,Y0)random
X=np.random.random(shape)
- sans initialisation des valeurs
# creation de tableaux
print(2*np.ones((3,2)))
print(np.arange(0.,1.,0.1))
print(np.linspace(0.,1.,11))
X,Y=np.meshgrid(np.linspace(0,1,3),np.linspace(0,1,2))
print("X=",X)
print("Y=",Y)
print("Valeurs aleatoires entre 0 et 1 =",np.random.random((5)))
initialisation avec des listes de compréhension
X = np.array([ val for i in range(n)])
print(np.array([i*i for i in range(10)]))
Indicage des tableaux¶
on compte à partir de 0 (comme en algorithmique)
X[0+i] élément i+1 de i
X[-1] dernier element de i = X[n-1]
X[-1-j] j élément avant le dernier = X[n-j]
X[i:j:pas] coupe ou slice du tableau X[i],X[i+pas],X[i+2p], .. X[k] avec k<j
X=np.array([j**3 for j in range(10)])
print(X)
print(X[0],X[1],X[2],X[-3],X[-2],X[-1])
print(X[-2:-1])
Adressage indirecte (fancy adressing)¶
on utilise des tableaux d’indices rol et col
A[row,col]A = np.array([[n+m*10 for n in range(5)] for m in range(5)])
print("A=",A)
row_indices=[0,2]
print("[A[i=0,2,:]]=\n",A[row_indices])
col_indices = [1, -1]
print("[A[0,1],A[2,-1]]=",A[row_indices,col_indices])
- utilisation de la fonction np.ix_ pour sélectionner des sous-matrices
print("sous matrice= ",A[np.ix_(row_indices,col_indices)])
- masque avec des valeurs logiques (bool)
- utile pour sélectionner les valeurs d’un vecteur dans un intervalle
B = np.linspace(0.,1.,5)
print("B=",B)
mask = np.array([True, False, False,True, True])
print(B[mask])
mask=(B>0.2)*(B<0.8)
print(B[mask])
Aliasing¶
print("pas d'aliasing avec les scalaires")
x=2
y=x
x=3
print(x,"#",y)
print("mais aliasing avec les listes et tableaux")
X=[1,2,3]
Y=X
X[0]=0
print(X,"==",Y)
utilisation de la fonction copy (explicite ou implicite)¶
print("pas d'aliasing si copie explicite des valeurs")
X=np.array([1,2,3])
Y=X.copy()
X[0]=0
print(X,Y)
Y=np.zeros(X.shape)
Y[:]=X
X[0]=3
print(X,Y)
Notation matricielle¶
- Attention par defaut opération sur les éléments
- les fonctions mathématiques dans numpy ont pour aguments des tableaux
- np.dot produit scalaire np.outer produit tensorielle np.inner
X=np.arange(0,3,1)
print("cos(X)=",np.cos(X))
print("X*X=",X*X)
print("X.X=",np.dot(X,X))
print("XoX=\n",np.outer(X,X))
print("XiX=",np.inner(X,X))
A=np.array(np.outer(np.arange(3),np.arange(3)))
print(type(A),A.shape)
print("A*X=\n",A*X)
print("A.X=\n",np.dot(A,X))
print("AoX=\n",np.outer(A,X))
print("AiA=\n",np.inner(A,A))
print("A.At=\n",np.dot(A,A.transpose()))
attention:¶
- on évite les boucles explicites pour des questions d’efficacité
- mais on commence toujours par écrire la forme explicite avec des boucles , puis ensuite la forme vectorielle
exemple: calcul de la forme quadratique $$ \Phi = \sum_{i=1}^n \sum_{j=1}^n A_{ij} X_i X_j - \sum_{j=1}^n B_j X_j$$
# calcul explicite
Phi=0.0
n=X.size
B=np.ones(X.shape)
for i in range(n):
sum=0.0
for j in range(n):
sum = sum + A[i,j]*X[j]
Phi = Phi + (sum - B[i])*X[i]
print(Phi)
# fonction numpy
print(np.dot(np.dot(A,X)-B,X))
calcul du résidu d’un système linéaire¶
$$R = B - A X$$$$R_i = B_i - \sum_{i=1}^n A_{ij} X_j$$ATTENTION: problème dans le code suivant.
trouver l'erreur!!!!!!!
n=5
B=np.random.random((n))
A=np.random.random((n,n))
X=np.random.random((n))
# calcul avec notation matricielle
R = B
R -= np.dot(A,X)
print("|R|=",np.linalg.norm(R))
# 2ne version
R = B - np.dot(A,X)
print("|R|=",np.linalg.norm(R))
# calcul explicite
R=np.zeros((n))
for i in range(n):
R[i]=B[i]
for j in range(n):
R[i] = R[i] - A[i,j]*X[j]
print("|R|=",np.linalg.norm(R))
erreur: problème aliasing
R[:]=BAnalyse de données d’un tableau¶
np.amax(tab) max suivant un axe
np.amin(tab) min
np.mean(tab) moyenne
np.sum(tab) somme
np.prod(tab) produitM=np.random.random((3,3))
print("M=",M)
print("max=",np.amax(M))
print("min=",np.amin(M,axis=1))
print("moy=",np.mean(M,axis=0))
print("sum=",np.sum(M))
print("prod=",np.prod(M,axis=0))
Lecture / Ecriture des données¶
gestion de fichiers classique avec Python¶
F=open("nom fichier","r")
L=F.readlines()
# decodage de chaque ligne
F.close()
lecture de données (format texte en colonnes)¶
A=np.loadtxt("nom de fichier")
ecriture d’un tableau de données¶
np.savetxt("nom de fichier",A)
ATTENTION utilisation de savetxt avec une variable fichier (File Handle):
- ouverture avec ‘wb’ (binary write)
Exemple: lecture fichier de données¶
%%bash
cat data/donnee.dat
# methode classique de lecture
F=open("data/donnee.dat","r")
L=F.readlines()
F.close()
# traitement des donnees
N=len(L)-1
X=np.zeros((N))
Y=np.zeros((N))
for i in range(N):
line = L[i+1].split()
X[i] = float(line[0])
Y[i] = float(line[1])
print("X=",X)
print("Y=",Y)
# version avec loadtxt
A=np.loadtxt("data/donnee.dat")
X=A[:,0]
Y=A[:,1]
print("X=",X)
print("Y=",Y)
Exemple: écriture fichier de données¶
A=np.random.rand(5,4)
F=open("data/data1.dat","w")
F.write("# fichier de resultat\n")
for i in range(A.shape[0]):
for j in range(A.shape[1]):
F.write("{0:8.4f} ".format(A[i,j]))
F.write("\n")
F.close()
# version avec savetxt
F=open("data/data2.dat","wb")
F.write("# fichier de resultat\n".encode('utf-8'))
np.savetxt(F,A)
F.close()
%%bash
cat data/data1.dat
cat data/data2.dat
Affichage avec un format ( printf en C)¶
print "format "%(val1,val2)
dans la chaine format : %d (decimal) %f (flottant) %s (chaine)
avec python 3:
print("x={},y={}".format(val1,val2))
print("x={1:fmt},y={0:fmt}".format(val2,val1))
avec pos (position) p.e. 0 1 et fmt (format) p.e. 8g 8f 8e
print("Pi=",np.pi)
print("Valeur approchée de Pi=%5.3f"%np.pi)
print("Valeur approchée de Pi={:5.2f}".format(np.pi))
Bibliothèque SCIPY¶
La bibliothéque SciPy (Scientifique Python) est basée sur Numpy et sa gestion efficace des tableaux multi-dimensionnels. Elle comprends entre autre des algorithmes pour:
- Fonctions mathématiques spéciales (scipy.special)
- Integration (scipy.integrate)
- Optimization (scipy.optimize)
- Interpolation (scipy.interpolate)
- Fourier Transforms (scipy.fftpack)
- Signal Processing (scipy.signal)
- Linear Algebra (scipy.linalg)
- Sparse Eigenvalue Problems (scipy.sparse)
- Statistics (scipy.stats)
- Multi-dimensional image processing (scipy.ndimage)
- File IO (scipy.io)
Fonctions spéciales¶
Fonctions de Bessel¶
Equivalent des fonctions trigonométriques sinus et cosinus en coordonnéees polaires
# fonction de Bessel de 1ere especes
from scipy.special import jn, jn_zeros
X=np.linspace(0,10,100)
plt.plot(X,jn(0,X),label="$J_0$")
plt.plot(X,jn(1,X),label="$J_1$")
plt.plot(X,jn(2,X),label="$J_2$")
plt.legend(loc=0)
print("Zeros de Jn(1)=",jn_zeros(1,5))
Polynômes (Chebyshev, Legendre, Laguerre, Hermite,..)¶
import numpy.polynomial.chebyshev as chebIntégration¶
Quadrature: calcul d’une valeur approchée¶
$$ I = \int_a^b f(x) dx$$from scipy.integrate import quad
def f(x):
return np.cos(x)
Ih,err=quad(f,0,np.pi/2)
print("Integrale =",Ih," Erreur =",err)
équations différentielles ordinaires (ODE)¶
Forme standard vectorielle avec $Y=[y_1,y_2,…y_n]$ avec une CI: $Y(0)=Y_0$ $$ Y’ = F(Y,t) $$
Pour une ODE d’ordre n, changement de variables: $$ y_1 = y, y_2 =y’,… y_n = y^{(n-1)}$$
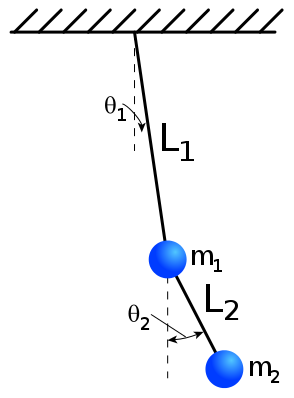
- Equation du mouvement (2 ddl, EDO d’ordre 2)
cas $L_1=L_2=l$ et $m_1=m_2=m$
Lagrangien 2 ddl
$$\begin{eqnarray}L(\theta_1,\theta_2,\dot{\theta_1},\dot{\theta_2},t) &=& \frac{ml^2}{6}[\dot{\theta_2}^2+4\dot{\theta_1}^2+3\dot{\theta_1}\dot{\theta_2}\cos(\theta_1-\theta_2)]\& + &\frac{mgl}{2}(3\cos\theta_1+\cos\theta2))\end{eqnarray}$$En posant $p_{\theta_1} =\frac{\partial L}{\partial \theta_1}$ et $p_{\theta_2} =\frac{\partial L}{\partial \theta_2}$ les quantités de mouvement généralisées, les équations de Lagrange s’écrivent
${\dot p_{\theta_1}} = -\frac{1}{2} m \ell^2 \left [ {\dot \theta_1} {\dot \theta_2} \sin (\theta_1-\theta_2) + 3 \frac{g}{\ell} \sin \theta_1 \right ]$
${\dot p_{\theta_2}} = -\frac{1}{2} m \ell^2 \left [ -{\dot \theta_1} {\dot \theta_2} \sin (\theta_1-\theta_2) + \frac{g}{\ell} \sin \theta_2 \right]$
En utilisant la définition de $p_{\theta_1}$ et $p_{\theta_2}$
${\dot \theta_1} = \frac{6}{m\ell^2} \frac{ 2 p_{\theta_1} - 3 \cos(\theta_1-\theta_2) p_{\theta_2}}{16 - 9 \cos^2(\theta_1-\theta_2)}$
${\dot \theta_2} = \frac{6}{m\ell^2} \frac{ 8 p_{\theta_2} - 3 \cos(\theta_1-\theta_2) p_{\theta_1}}{16 - 9 \cos^2(\theta_1-\theta_2)}.$
- Changement de variable (4 ddl, 4 EDO d’ordre 1) $$x = [x_1=\theta_1, x_2=\theta_2, x_3=p_{\theta_1}, x_4=p_{\theta_2}]$$
${\dot x_1} = \frac{6}{m\ell^2} \frac{ 2 x_3 - 3 \cos(x_1-x_2) x_4}{16 - 9 \cos^2(x_1-x_2)}$
${\dot x_2} = \frac{6}{m\ell^2} \frac{ 8 x_4 - 3 \cos(x_1-x_2) x_3}{16 - 9 \cos^2(x_1-x_2)}$
${\dot x_3} = -\frac{1}{2} m \ell^2 \left [ {\dot x_1} {\dot x_2} \sin (x_1-x_2) + 3 \frac{g}{\ell} \sin x_1 \right ]$
${\dot x_4} = -\frac{1}{2} m \ell^2 \left [ -{\dot x_1} {\dot x_2} \sin (x_1-x_2) + \frac{g}{\ell} \sin x_2 \right]$
g = 9.82
L = 0.5
m = 0.1
def F(x, t):
""" 2nd membre F(X) des équations ODE du pendule double """
x1, x2, x3, x4 = x[0], x[1], x[2], x[3]
dx1 = 6.0/(m*L**2) * (2 * x3 - 3 * np.cos(x1-x2) * x4)/(16 - 9 * np.cos(x1-x2)**2)
dx2 = 6.0/(m*L**2) * (8 * x4 - 3 * np.cos(x1-x2) * x3)/(16 - 9 * np.cos(x1-x2)**2)
dx3 = -0.5 * m * L**2 * ( dx1 * dx2 * np.sin(x1-x2) + 3 * (g/L) * np.sin(x1))
dx4 = -0.5 * m * L**2 * (-dx1 * dx2 * np.sin(x1-x2) + (g/L) * np.sin(x2))
return [dx1, dx2, dx3, dx4]
def Energie(x):
""" calcul de l'energie totale du système """
t1, t2, p1, p2 = x[0], x[1], x[2], x[3]
dt1 = 6.0/(m*L**2) * (2 * p1 - 3 * np.cos(t1-t2) * p2)/(16 - 9 * np.cos(t1-t2)**2)
dt2 = 6.0/(m*L**2) * (8 * p2 - 3 * np.cos(t1-t2) * p1)/(16 - 9 * np.cos(t1-t2)**2)
Et = L**2/6*(4*dt1**2+dt2**2+3*dt1*dt2*np.cos(t1-t2)) - g*L/2*(3*np.cos(t1)+np.cos(t2))
return Et
from scipy.integrate import odeint
# cdts initiale: cas petites oscillations
x0 = [np.pi/20, np.pi/20, 0, 0]
# calcul de la solution en n points sur l'intervalle 0 10s
t = np.linspace(0, 10, 1000)
# integration numérique
x = odeint(F, x0, t, rtol=1.e-8,atol=1.e-10)
# coordonnees du pendule
x1 = + L * np.sin(x[:, 0])
y1 = - L * np.cos(x[:, 0])
x2 = x1 + L * np.sin(x[:, 1])
y2 = y1 - L * np.cos(x[:, 1])
# calcul energie totale
Et = [Energie(xi) for xi in x]
# tracer de la solution
plt.figure(figsize=(12,6))
plt.subplot(1,2,1)
plt.plot(t,x[:,0],lw=2,label="$\\theta_1$")
plt.plot(t,x[:,1],lw=2,label="$\\theta_1$")
plt.legend(loc=0)
plt.subplot(1,2,2)
plt.plot(x1,y1)
plt.plot(x2,y2)
plt.axis('equal')
plt.title('trajectoire')
# verification
plt.figure(figsize=(10,6))
plt.plot(t,Et,lw=2)
plt.title("E. totale")
Transformation de Fourier¶
Analyse du signal $\theta_1$ et $\theta_2$ précédent pour mettre en évidence les 2 modes propres du système. Comparaison avec la fréquence du pendule simple.
from scipy.fftpack import fft,fftfreq
# FFT de theta1 et theta2
n2 = x.shape[0]//2
f1 = fft(x[:,0])
f2 = fft(x[:,1])
# plage de frequence
dt=t[1]-t[0]
w = fftfreq(len(t), dt)
# frequence de base
F0 = np.sqrt(g/L)/(2*np.pi)
plt.title("spectre")
plt.semilogy(w[:n2],abs(f1[:n2]),lw=2,label="$\\theta_1$")
plt.semilogy(w[:n2],abs(f2[:n2]),lw=2,label="$\\theta_2$")
plt.xlim(0,10)
plt.semilogy([F0,F0],[0.01,100.],'--k')
plt.legend()
Algèbre linéaire¶
Résolution système linéaire $A x = b$¶
import scipy.linalg as lin
lin.solve(A,B) résolution AX=B
lin.inv(A) calcul inverse de A !!!
lin.det(A) déterminant de A
lin.norm(A,ord=n) norme de A
A.conjugate()
A.transpose()
A.real
A.imag
M.max(axis=0)
M.min()
np.diag(A)
np.trace(A)n=2
A=np.random.random((n,n))
M = A + 1j*A.transpose()
print("conjugué\n",M.conjugate())
print("transpose (attention #)\n",M.transpose())
print("parti imaginaire\n",M.imag)
print("Diag A=",np.diag(A))
print("trace A=",np.trace(A))
import scipy.linalg as lin
A = np.array([[9,2,3], [4,9,6], [7,8,9]],dtype=float)
b = np.array([1,2,3],dtype=float)
print("det A=",lin.det(A))
x = lin.solve(A,b)
r = b - np.dot(A,x)
print("erreur =",lin.norm(r))
Problème aux valeurs propres¶
- calcul valeurs propres $\lambda_n$ et vecteurs propres $u_n$ t.q. : $$A u_n = \lambda_n u_n $$
evals = lin.eigvals(A)
print("Valeurs propres \n",evals)
print("determinant de A=",lin.det(A))
evals,evects = lin.eig(A)
print("vecteurs propres:\n",evects)
A = np.array([[4,-1,-1], [-1,4,-1], [-1,-1,4]],dtype=float)
print("vp de A=",lin.eigvals(A))
print("norme l2=",lin.norm(A,ord=2))
Interpolation¶
Interpolation polynomiale $p(x)$ de $f(x)$ passant par $n$ points $x_i$, i.e. tq $p(x_i)=f(x_i)$
import scipy.interpolate as interpimport scipy.interpolate as interp
def f(x):
return np.cos(x)
# pts d'interpolation
n=10
xp = np.linspace(0,10.0,n)
yp = f(xp)
X = np.linspace(0,10.0,100)
p1 = interp.interp1d(xp, yp)
p3 = interp.interp1d(xp, yp, kind='cubic')
plt.figure(figsize=(12,6))
plt.subplot(1,2,1)
plt.plot(xp,yp,'o')
plt.plot(X,f(X),'-k')
plt.plot(X,p1(X),'-',lw=2,label='lineaire')
plt.plot(X,p3(X),'-',lw=2,label='cubique')
plt.legend(loc=0)
plt.subplot(1,2,2)
plt.plot(X,p1(X)-f(X),'-',lw=2,label='lineaire')
plt.plot(X,p3(X)-f(X),'-',lw=2,label='cubique')
plt.legend(loc=0)
plt.title('Erreur')
Tracé avec Matplotlib¶
Matplotlib est une execellente bibliothèque Python pour du graphique 2D et 3D de données scientifiques.
- Facile à utiliser
- Support de $\LaTeX$
- Contrôle de chaque élément d’une figure, y compris la taille de la figure et son DPI.
- Sortie de haute qualité dans de nombreux formats, y compris PNG, PDF, SVG, EPS et PGF.
- GUI et travail en mode batch
Une des caractéristiques de Matplotlib est le contrôle complet de la figure par le programmeur. C’est un point important pour la reproductibilité des résultats lorsque l’on a besoin de régénérer la figure avec des données mises à jour.
Plus d’informations sur la page Web Matplotlib: http://matplotlib.org/
Utilisation sous Python
import matplotlib.pyplot as plt# donnees
F = lambda x: np.sin(x)/x
x = np.linspace(1.e-10, 10, 100)
y = F(x)
# tracer
fig = plt.figure(figsize=(8,8))
axes1 = fig.add_axes([0.1, 0.1, 0.8, 0.8]) # main axes
axes2 = fig.add_axes([0.4, 0.5, 0.4, 0.3]) # inset axes
# main figure
axes1.plot(x, y,'--b',lw=2)
axes1.set_xlabel('x')
axes1.set_ylabel('y')
axes1.axhline(y=0,c='k')
axes1.set_title('fonction $y=f(x)$')
# insert
axes2.plot(y, x, 'g',lw=2)
axes2.set_xlabel('$y=\\frac{sin(x)}{x}$')
axes2.set_ylabel('x')
axes2.set_title('fonction inverse $y=f^{-1}(x)$');
Exemples sur le site de matplotlib¶
from IPython.display import HTML
HTML('<iframe src=http://matplotlib.org/gallery.html width=700 height=350></iframe>')
Documentations supplémentaires¶
- http://www.scipy.org - The official web page for the SciPy project.
- http://docs.scipy.org/doc/scipy/reference/tutorial/index.html - A tutorial on how to get started using SciPy.
- http://numpy.scipy.org librairie scipy & numpy
- http://scipy.org/Tentative_NumPy_Tutorial
- http://scipy.org/NumPy_for_Matlab_Users - A Numpy guide for MATLAB users.
Version de Python et des bibliothéques¶
print("\t\tSystème utilisé")
import sys
print("Système :\t\t",sys.platform)
import platform
print(platform.platform())
print("Ordinateur:\t\t",platform.machine())
print("Version de Python:\t",sys.version)
import IPython
print("Version de IPython:\t",IPython.__version__)
import numpy
print("Version de numpy:\t",numpy.version.version)
import scipy
print("Version de scipy:\t",scipy.version.version)
import matplotlib
print("Version de matplotlib:\t",matplotlib.__version__)